An orthogonal T7 replisome for continuous hypermutation and accelerated evolution in E. coli (T7-ORACLE)
Introducción
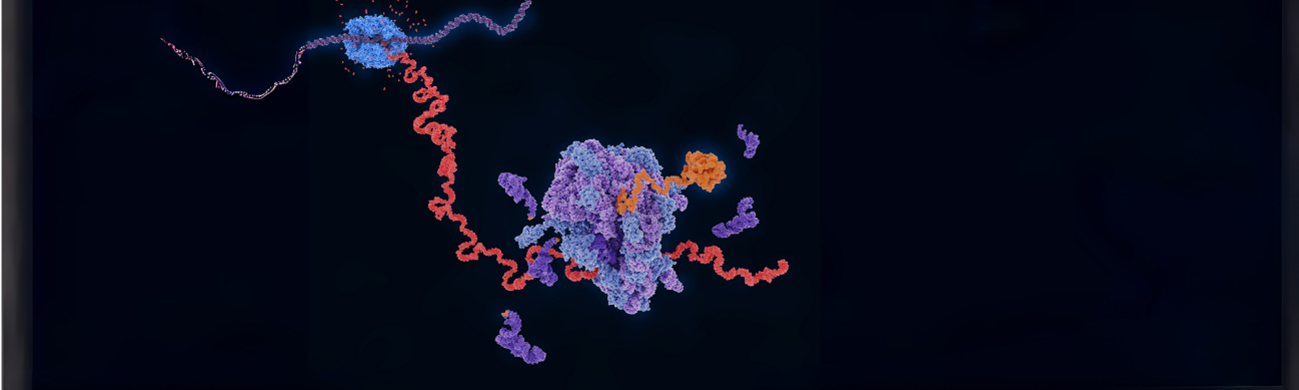
El trabajo publicado en Science en 2025 presenta T7-ORACLE, un sistema de replicación ortogonal diseñado para inducir hipermutación continua en genes específicos dentro de Escherichia coli. La evolución dirigida de proteínas constituye una de las estrategias más potentes para obtener variantes con nuevas funciones, pero los métodos clásicos se ven limitados por ciclos de mutagénesis y selección que requieren múltiples pasos manuales y tiempos de ejecución prolongados. Para superar estas limitaciones, los autores desarrollaron un replisoma derivado del bacteriófago T7 capaz de replicar plásmidos circulares de forma independiente al genoma bacteriano, incorporando una ADN-polimerasa T7 de baja fidelidad. Esta polimerasa hipermutagénica introduce mutaciones con gran frecuencia exclusivamente en los plásmidos, evitando comprometer la estabilidad del ADN cromosómico de la bacteria. El objetivo del estudio fue demostrar que T7-ORACLE permite un proceso de evolución continua, rápida y dirigida en un sistema bacteriano estándar, validando su eficacia con el modelo clásico de la β-lactamasa TEM-1, una enzima de resistencia a antibióticos.
Resultados
La ingeniería del sistema comenzó con la introducción controlada del replisoma T7 en E. coli, configurado para replicar únicamente plásmidos ortogonales. Las variantes mutagénicas de la polimerasa T7 alcanzaron tasas de mutación de aproximadamente 1,7 × 10⁻⁵ sustituciones por base in vivo, lo que supone un incremento cercano a 100 000 veces respecto a la tasa basal de mutación en el genoma bacteriano. De esta manera, los autores generaron un microentorno altamente dinámico en el que los genes clonados en los plásmidos evolucionaban de manera continua en paralelo al crecimiento bacteriano. Como prueba de concepto, se utilizó el gen de la β-lactamasa TEM-1, responsable de conferir resistencia a antibióticos betalactámicos. Cepas transformadas con plásmidos que contenían dicho gen fueron sometidas a presiones selectivas crecientes con antibióticos clínicamente relevantes, entre ellos monobactámicos y cefalosporinas. En menos de una semana de cultivo y selección, emergieron variantes con mejoras drásticas en su actividad catalítica. Algunas de estas variantes mostraron una actividad hasta 5000 veces superior contra antibióticos, con mutaciones puntuales que coincidían en parte con aquellas observadas en entornos clínicos reales. Además, se identificaron configuraciones mutacionales inéditas que superaron incluso los niveles de resistencia descritos previamente, lo que evidencia el potencial del sistema no solo para reproducir fenómenos clínicos de resistencia, sino también para descubrir soluciones evolutivas novedosas.
Conclusiones y discusión
El estudio demuestra que T7-ORACLE constituye una herramienta robusta para evolución dirigida acelerada, ya que combina una elevada tasa de mutagénesis focalizada con la preservación de la estabilidad cromosómica del hospedador. Al estar restringida a plásmidos, la hipermutación evita efectos deletéreos en el metabolismo bacteriano y permite mantener cultivos viables en condiciones de selección intensa. El carácter ortogonal del replisoma lo hace compatible con procedimientos estándar de biología molecular, ya que funciona con plásmidos circulares convencionales y no requiere equipamiento especializado, lo que facilita su adopción generalizada en laboratorios. A diferencia de los enfoques tradicionales que implican rondas discretas de mutagénesis y selección, T7-ORACLE genera mutaciones de manera continua en cada ciclo de replicación bacteriana, lo que reduce drásticamente los tiempos experimentales y permite obtener variantes funcionales en días en lugar de semanas o meses. La validación con la β-lactamasa TEM-1 ilustra de forma contundente su utilidad en el estudio de resistencia antibiótica, al reproducir patrones mutacionales clínicos y generar nuevas configuraciones con relevancia biomédica. Sin embargo, el potencial del sistema trasciende el ámbito de los antibióticos: genes de enzimas humanas, proteínas virales, anticuerpos o polimerasas artificiales podrían clonarse en plásmidos ortogonales y someterse al mismo proceso evolutivo. Así, T7-ORACLE abre una vía hacia la reprogramación rápida y dirigida de proteínas, con aplicaciones que abarcan la biomedicina, la biocatálisis, la biotecnología industrial y la ingeniería de enzimas con funciones novedosas. En conclusión, este sistema representa un avance decisivo en evolución dirigida en células vivas, capaz de transformar la forma en que se generan y seleccionan proteínas funcionales a escala experimental.
Referencia:
Diercks C.S., Sondermann P.J., Rong C., Dik D.A., Gillis T.G., Ban Y., Schultz P.G. et al. (2025). An orthogonal T7 replisome for continuous hypermutation and accelerated evolution in E. coli. Science. 389(6760):618–622.https://pubmed.ncbi.nlm.nih.gov/39211285/